La découverte
En 1989, Francis MOJICA, un chercheur d’Alicante, s’intéresse à Haloferax Mediterranei (un organisme unicellulaire) pour comprendre sa résistance aux fortes concentrations en sel dans les marais et trouve une séquence d’ADN qui se répète sans comprendre son utilité, ce qui l’intrigue. Il s’aperçoit que cette même séquence apparaît chez les bactéries E. coli et s’intéresse alors plus spécifiquement à cette bactérie.
MOJICA va échanger avec d’autres chercheurs qui travaillent aussi sur le même sujet dont Ruud Jansen et en 2002, apparaît pour la première fois le nom de CRISPR dans une publication de Jansen1 sans que l’on ne comprenne encore son fonctionnement. En août 2002, MOJICA comprend que ce qu’il étudie est un système immunitaire adaptatif chez les bactéries qui intègrent dans leur génome de l’ADN des virus agresseur pour les repérer plus facilement et, à l’avenir, effectuer des attaques préventives en coupant l’ADN des virus bactériophages.
Mais MOJICA ne réussit pas à immédiatement publier ses découvertes car la plupart des rédacteurs n’arrivaient pas à croire en ce que disait MOJICA. Il finit par publier son article le 1er février 2005 dans Journal of Molecular Evolution.Il faut attendre février 2007 et l’équipe de Danisco, une société de biotechnologie qui travaille dans la production alimentaire, pour démontrer expérimentalement qu’il s’agit bien d’un système immunitaire adaptatif c’est-à-dire un système qui mémorise l’ADN des virus qu’il a rencontré, pour s’en prémunir lors de nouvelles attaques en le repérant plus rapidement.
Crédit photo: Francceinter (2017, 26 janvier)3
En 2009, Jennifer Doudna de Berkeley et Emmanuelle Charpentier de l’institut Max Planck de Berlin commencent leurs recherches sur CRISPR dans le but de guérir des maladies génétiques. En effet, en inactivant certains gènes responsables de maladies génétique de la même manière que la bactérie inactive l’ADN du virus bactériophage, elles espèrent pouvoir apporter avec CRISPR-Cas9 des thérapies géniques moins chers et plus facilement mise en place. C’est en 2012 que CRISPR-Cas9 est révélé au grand jour par Jennifer Doudna et Emmanuelle Charpentier dans un article de Science2 décrivant le mécanisme de la technique CRISPR-Cas9, une technique de modification de l’ADN révolutionnaire.
Cette publication est le point de recherches intenses sur le sujet des CRISPR comme l’attestent les statistiques de publication traitant de CRISPR.
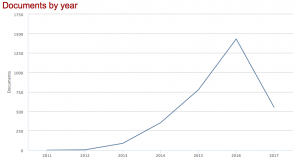
Nombre d’articles scientifiques traitant la technique CRISPR-Cas9 parus dans le monde d’après les données de la plateforme Scopus (réalisé par Badis Marsit)
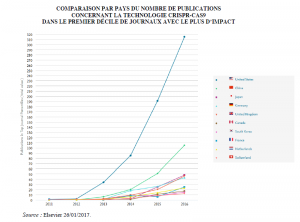 Rapport de l’OPECST
Rapport de l’OPECST
Les différentes méthodes CRISPR
Les méthodes de modification CRISPR, comme les autres nouvelles techniques de modification génétique et de croisement végétal (New Plant Breeding Technology -NPBT) agissent localement sur l’ADN en repérant des séquences d’ADN. C’est ce que l’on appelle en anglais une modification Site Directed Nucleases ou SDN, on parle de nucléases ciblées. Les SDN ne sont pas propres à CRISPR-Cas9 mais nous allons principalement nous concentrer sur CRISPR-Cas9.
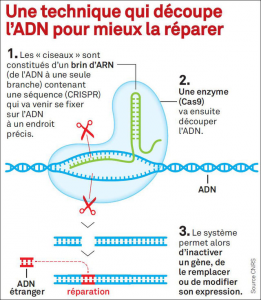
On peut compter 3 types de modifications SDN. Ces trois techniques se fondent sur une cassure de la double hélice d’ADN (Double Strand Break – DSB- dans la littérature internationale) de la cellule cible. Dans le cas de CRISPR-Cas9, c’est l’enzyme Cas9 qui coupe et les séquences CRISPR guident cette cassure. Un mécanisme de réparation de l’ADN ciblé par la cassure est alors déclenché dans le noyau mais ce dernier produit une rémission aléatoire. Cela génère souvent des mutations (insertion, délétion ou substitution nucléotidique) ciblées sur la zone de la cassure.
Le caractère aléatoire de la réparation de l’ADN après la cassure fait parfois dire que la méthode CRISPR comme toutes les NBT-New Breeding Technology au sens large- sont des “mutagenèses ciblées”. C’est-à-dire que le but de la manipulation est de provoquer des mutations par des coupures d’ADN ciblées grâce à l’association d’un CRISPR et d’une enzyme Cas9.
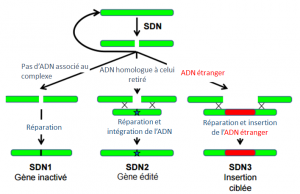 Schéma inspiré du schéma du rapport des Rencontres Agriculture & Innovation 2025: “promesses et limites des nouveaux outils de sélection végétale”
Schéma inspiré du schéma du rapport des Rencontres Agriculture & Innovation 2025: “promesses et limites des nouveaux outils de sélection végétale”
SDN 1 : L’inactivation
Une modification de type SDN-1 se produit lorsqu’aucun d’ADN n’est associé au complexe CRISPR-Cas9. Le produit de cette modification sur le brin d’ADN est une inactivation ciblée d’une portion d’ADN. La localisation de la DSB (double strand break, cassure double-brin) est ciblée par la nucléase Cas9 et la reconstruction de l’ADN a posteriori peut générer des délétions, des substitutions ou des insertions.
Si deux cassures double brins sont opérées de part et d’autre d’une séquence d’ADN ciblée, une SDN-1 peut résulter dans la délétion d’une large bande de nucléotides.
Parmi les projets en cours, CRISPR est utilisé en SDN-1 pour augmenter la résistance au mildiou chez le blé4 en inactivant trois occurrences du génome qui rendait le blé vulnérable au mildiou.
SDN 2 : L’édition de gènes
Une SDN-2 est foncièrement différente d’une SDN-1 même si les effets sont proches.
En effet une SDN-2 génère une cassure mais cette fois, la réparation est guidée par un ADN modèle qui est majoritairement identique à la séquence sélectionnée sauf quelques modifications ciblées (de l’ordre de quelques nucléotides). Ces modifications seront induites sur l’hôte modifiant ces quelques nucléotides : il ne s’agit donc pas d’ADN d’une autre espèce.
Le produit d’une SDN-2 est donc une mutation ciblée sur un gène déterminé, on notera l’exemple de la résistance à l’herbicide BS (bispyribac sodium) chez le riz5.
SDN 3 : Insertion ciblée
La technique de modification SDN-3 permet l’insertion ciblée d’un gène d’une autre espèce.Comme pour les autres SDN, une DSB est opérée. La réparation est cette fois guidée par un modèle contenant des séquences étrangères d’ADN. Il faut cependant distinguer : la cisgénèse (la mutation pourrait être obtenue par des techniques d’hybridation classiques), et la transgénèse (les gènes proviennent d’espèces non apparentées).
Comme une DSB génère une réparation ponctuelle, ces techniques ( et surtout la technique SDN 1) sont, en théorie, observables sans intervention humaine. Ce qui les rend de facto naturelles. Cette vision est contestée par certains experts de la traçabilité comme Yves Bertheau qui y voit une négligence du stress de la manipulation sur la cellule qui rend celle-ci irrémédiablement artificielle. En effet, pour introduire le brin d’ARN CRISPR guide et l’enzyme Cas9, on utilise des vecteurs viraux ou on encapsule le complexe dans un plasmide. Cela laisse des cicatrices sur les membranes de la cellule et de son noyau. Corrélées avec d’autres indices, ces cicatrices montreraient que la cellule a bien subie une manipulation artificielle. Pour en savoir plus
[1] Jansen R, Embden J., Gaastra W, Schouls L. (2002, Mars) Identification of genes that are associated with DNA repeats in prokaryotes. Molecular microbiology
[2] Herzberg N. (2016 , 28 juillet) Crispr, le big bang de la génétique La saga Crispr – Cas9 1|6. Le Monde
[3] Jost T. (2017, 26 janvier). Crispr Cas9 : la grande menace [Vidéo en ligne] [1’15’’]. Disponible sur : https://www.franceinter.fr/sciences/crispr-cas9-la-grande-menace . [Consulté le 09/06/2017]
[4] Talbot T. (2014, 21 juillet) Chinese Researchers stop Wheat Disease with Gene Editing, MIT Technology Review. Disponible sur https://www.technologyreview.com/s/529181/chinese-researchers-stop-wheat-disease-with-gene-editing/
[5] Sun Y. ,Zhang X., Wu C., He Y., Ma Y., Hou H., Guo X., Du W., Zhao Y., Xia L. (2016, 5 janvier) Engineering Herbicide-Resistant Rice Plants through CRISPR/Cas9-Mediated Homologous Recombination of Acetolactate Synthase. Molecular Plant. Disponible sur http://www.cell.com/molecular-plant/abstract/S1674-2052(16)00002-2